一个重要的闭环:
机器学习-数据挖掘的流程(CRISP-DM):围绕数据进行如下6个活动进行闭环式地探索活动
- 商业理解
- 数据理解
- 数据准备
- 建立模型
- 模型评估
- 方案实施
一个重要的概念:
特征工程:最大限度地从原始数据中提取特征以供算法和模型使用,包括如下几个主要部分:
- 数据预处理:标准化、缩放、缺失、变换、编码等
- 特征产生:结合业务数据、派生新的特征
- 特征选择:通过各种统计量、模型评分等,筛选合适的特征
- 降维:PCA、LDA等减少特征个数
两个重要的算法:
决策树:
- 构建决策树
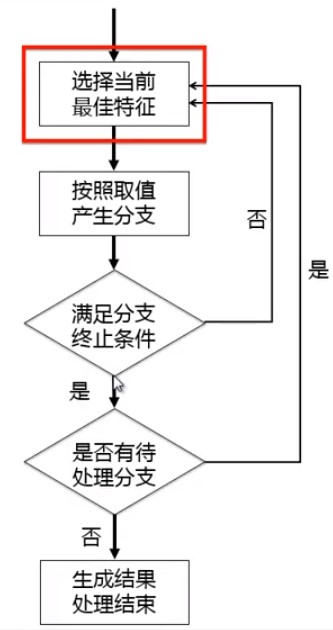
- 如何“选择当前最佳特征”:

其中包含的主要概念:
信息熵:信息论理的概念,香农提出;描述混乱程度的度量;取值范围:0~1(值越大,越混乱)。计算公式如下:

信息增益:信息是确定性的增加;从一个状态到另一个状态信息的变化;信息增益越大,对确定性贡献越大。
- 决策树算法的主要分类:
- ID3系列(Iterative Dichotomiser 3, 迭代树三代):核心是信息熵,根据信息增益决策树的节点;存在一些问题:信息度量不合理-倾向于选择取值多的字段;输入类型单一 - 离散型;不做剪枝,容易过拟合。
- C4.5:和ID3相比的改进:用信息增益率代替信息增益;能对连续属性进行离散化,对不完整数据进行处理;进行剪枝。
- C50:C4.5相比的改进:使用了boosting; 前修剪、后修剪
- CART(Classification and Regression Tree): 核心是基尼系数(Gini); 分类是二叉树;支持连续值和离散值;后剪枝进行修剪;支持回归,可以预测连续值
- 决策树的具体施行:
-
集成学习:针对同一数据集,训练多种学习器,来解决同一问题。
- Bagging: 有放回抽样构建多个子集;训练多个分类器;最终结果由各分类器结果投票得出;(实现非常简单)。

-
Boosting: 重复使用一类学习器来修改训练集;每次训练后根据结果调整样本的权重;每个学习器加权后的线性组合即为最终结果。
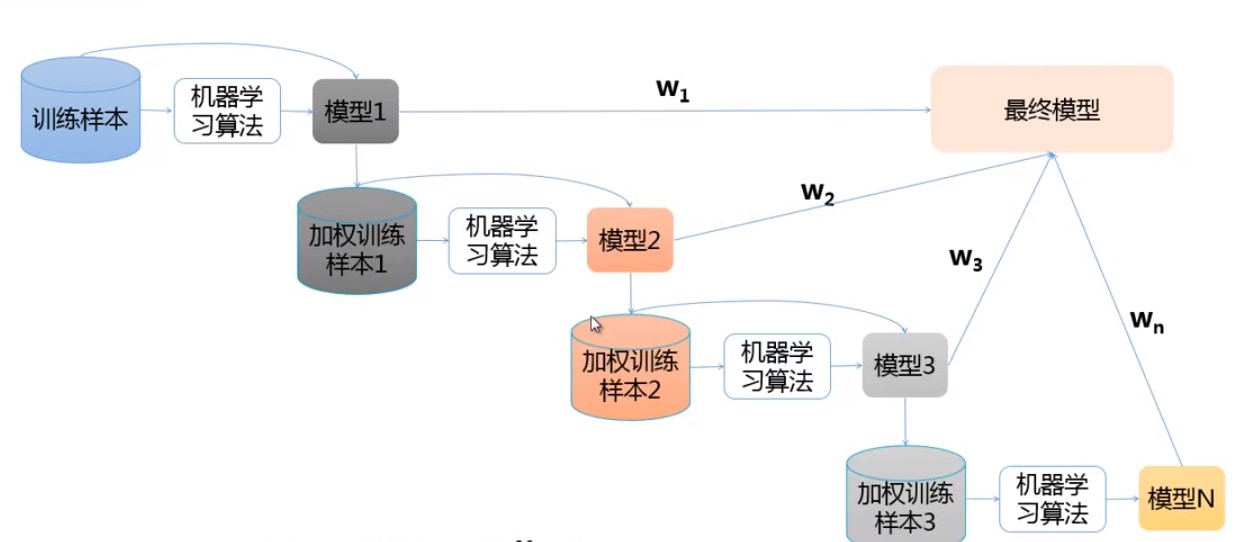
-
AdaBoost:
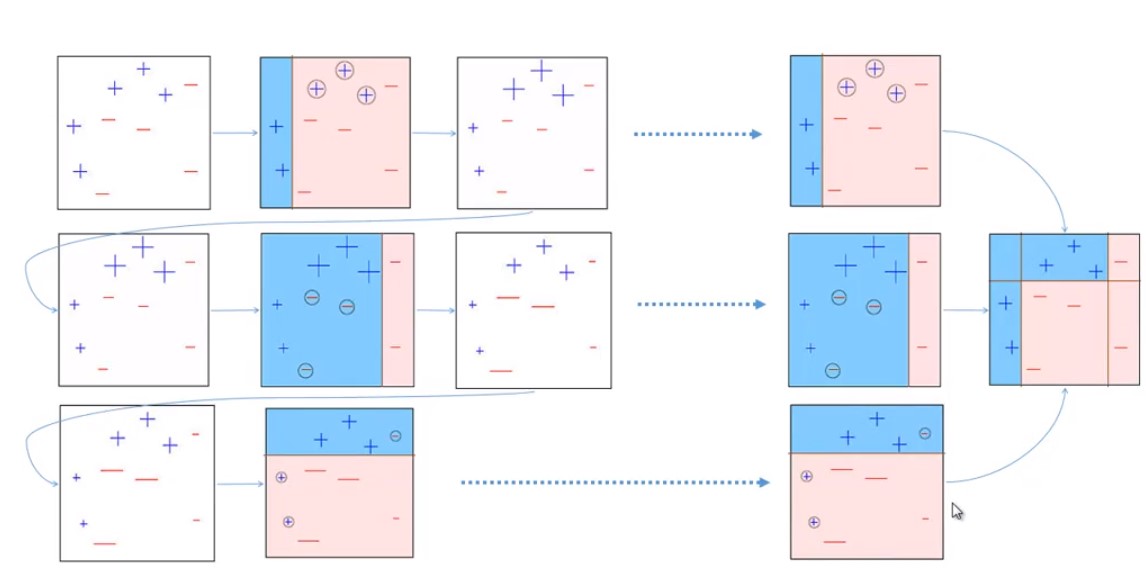
- 其他实现方法:
Stacking: 由两级组成,第一级为初级学习器,第二级为高级学习器;第一级学习器的输出作为第二级学习器的输入。
随机森林:
由许多决策树组成,树生成时采用了随机的方法;Smart Bagging;生成步骤:1.随机采样,生成多个样本集;2.对每个样本集构建决策树。
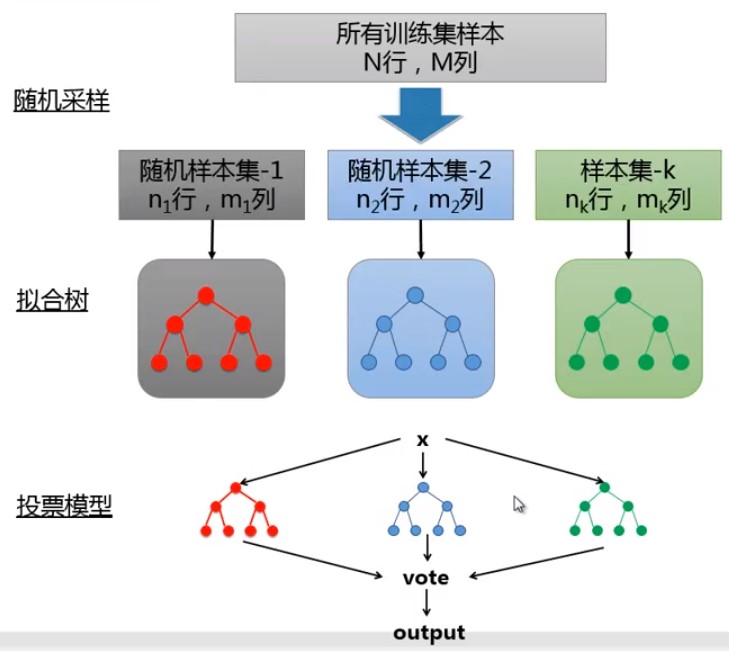
其优点包括:可以处理多分类;不会过拟合;容易实现并行;对数据集容错能力强。
参考 - 1. R语言运用随机森林的例子:
###################################################
### Gene (Feature) Selection 基因特征选择: 1.过滤方法;2.封装方法
###################################################
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install(version = "3.11")
BiocManager::install(c("Biobase", "genefilter"))
BiocManager::install(c("ALL"))
library(Biobase)
library(ALL)
data(ALL)
ALLb <- ALL[,tgt.cases]
rowIQRs <- function(em)
rowQ(em,ceiling(0.75*ncol(em))) - rowQ(em,floor(0.25*ncol(em)))
plot(rowMedians(es),rowIQRs(es),
xlab='Median expression level',
ylab='IQR expression level',
main='Main Characteristics of Genes Expression Levels')
library(genefilter)
ALLb <- nsFilter(ALLb,
var.func=IQR,
var.cutoff=IQR(as.vector(es))/5,
feature.exclude="^AFFX")
ALLb <- ALLb$eset
es <- exprs(ALLb)
dim(es)
#ANOVA过滤
f <- Anova(ALLb$mol.bio,p=0.01)
ff <- filterfun(f)
selGenes <- genefilter(exprs(ALLb),ff)
sum(selGenes)
ALLb <- ALLb[selGenes,]
ALLb
es <- exprs(ALLb)
plot(rowMedians(es),rowIQRs(es),
xlab='Median expression level',
ylab='IQR expression level',
main='Distribution Properties of the Selected Genes')
# 用随机森林(适合用于处理包含大量特征的问题)进行过滤:随机森林由一组决策树构成,取决于分析的问题采用回归树还是分类树 - 每棵树都是通过自助法抽样(从原始数据集中用有放回抽样法随机抽取N个个案)进行训练
# 对于回归问题,采用每棵树的预测值得平均值作为这些组合的预测值。对于分类问题,则采用投票机制
featureNames(ALLb) <- make.names(featureNames(ALLb))
es <- exprs(ALLb)
library(randomForest)
dt <- data.frame(t(es),Mut=ALLb$mol.bio)
rf <- randomForest(Mut ~ .,dt,importance=T)
imp <- importance(rf)
imp <- imp[,ncol(imp)-1]
rf.genes <- names(imp)[order(imp,decreasing=T)[1:30]]
sapply(rf.genes,function(g) tapply(dt[,g],dt$Mut,median))
library(lattice)
ordMut <- order(dt$Mut)
levelplot(as.matrix(dt[ordMut,rf.genes]),
aspect='fill', xlab='', ylab='',
scales=list(
x=list(
labels=c('+','-','*','|')[as.integer(dt$Mut[ordMut])],
cex=0.7,
tck=0)
),
main=paste(paste(c('"+"','"-"','"*"','"|"'),
levels(dt$Mut)
),
collapse='; '),
col.regions=colorRampPalette(c('white','orange','blue'))
)
#用特征聚类的组合进行过滤
library(Hmisc)
vc <- varclus(t(es))
clus30 <- cutree(vc$hclust,30)
table(clus30)
getVarsSet <- function(cluster,nvars=30,seed=NULL,verb=F)
{
if (!is.null(seed)) set.seed(seed)
cls <- cutree(cluster,nvars)
tots <- table(cls)
vars <- c()
vars <- sapply(1:nvars,function(clID)
{
if (!length(tots[clID])) stop('Empty cluster! (',clID,')')
x <- sample(1:tots[clID],1)
names(cls[cls==clID])[x]
})
if (verb) structure(vars,clusMemb=cls,clusTots=tots)
else vars
}
getVarsSet(vc$hclust)
2. R语言数据科学包列表 : https://www.cnblogs.com/yxmings/p/14213573.html
文章来源: 博客园
- 还没有人评论,欢迎说说您的想法!



 客服
客服


